地角天邊
江郎才盡,不足為訓
SAS編程技術教程(朱世武).zip_網盤鏈接頁麵 (sopandas.cn)
epdf.pub_applied-multivariate-statistical-analysis-6th-edit.pdf (archive.org)
統計學基本知識一 https://blog.csdn.net/fanzonghao/article/details/87929764
https://blog.csdn.net/qq_22592457/article/details/92982170
1、基礎
因子:分類自變量;協變量:連續型自變量。
1.1 有關平均值參數u的假設檢驗
根據是否已知方差,分為兩類檢驗:U檢驗和T檢驗。(當為μ即均值,σ是方差) 如果已知方差,則使用U檢驗,如果方差未知則采取T檢驗。【汪海波:當樣本含量 n 較大時,樣本均數符合正態分布,故可用 u 檢驗進行分析。當樣本含量 n 小時,若觀察值 x 符合正態分布,則用 t 檢驗(因此時樣本均數符合 t 分布),當 x 為未知分布時,則應采用秩和檢驗。Wilcoxon的秩和檢驗(rank sum test)不依賴於總體分布的具體形式。】
1.2、有關參數方差σ2的假設檢驗
F檢驗是對兩個正態分布的方差齊性檢驗,簡單來說,就是檢驗兩個分布的方差是否相等。【汪海波:t 檢驗和 u 檢驗不適用於多個樣本均數的比較,因多次比較置信度下降。R.A.Fisher 的方差分析(analysis of variance,ANOVA)以 F 命名其統計量,又名 F 檢驗。】
1.3、檢驗兩個或多個變量之間是否關聯
卡方檢驗(Karl Pearson 卡方檢驗 - 知乎 )屬於非參數檢驗,主要是比較兩個及兩個以上樣本率(構成比)以及兩個分類變量的關聯性分析。根本思想在於比較理論頻數和實際頻數的吻合程度或者擬合優度問題。https://blog.csdn.net/qq_22592457/article/details/92982170
1.4 Wilcoxon的秩和檢驗
Wilcoxon_rank_sum test: SAS instruction (purdue.edu)
1.5 線性混合效應模型(linear mixed effects model)
1.5.1 https://blog.csdn.net/weixin_43569478/article/details/108079707
Linear Mixde Model, 簡稱LMM, 稱之為線性混合模型。從名字也可以看出,這個模型和一般線性模型有著很深的淵源。線性混合模型是在一般線性模型的基礎上擴展而來,在回歸公式中同時包含了以下兩種效應
fixed-effects, 固定效應
random efffects,隨機效應
其名稱中的混合一詞正是來源於此。一元簡單線性模型的公式如下:
其中X代表固定效應,ε表示隨機誤差,而線性混合模型的公式如下:
相比簡單線性模型,多出了Z這一項,這一項稱之為隨機效應。當然兩種模型的本質並不是體現在回歸公式中自變量的多少,而在於自變量的類別,在一般線性模型中,其自變量全部為固定效應自變量,而線性混合模型中,除了固定效應自變量外,還包含了隨機效應自變量。
所以關鍵之處在於判定自變量的類別,如果一個自變量的所有類別在抽樣的數據集中全部包含,則將該變量作為固定效應,比如性別,隻要抽樣的數據中同時包含了兩種性別,就可以將性別作為固定效應自變量;如果一個自變量在抽樣的數據集中的結果隻是從總體中隨機抽樣的結果,那麽需要作為隨機效應自變量。簡而言之,如果抽樣數據集中的自變量可以包含該自變量的所有情況,則作為固定效應,如果隻能代表總體的一部分,則作為隨機效應。
在分析的時候,可以將自變量都作為固定效應自變量,然後用一般線性模型來進行處理,那麽為何要引入隨機效應自變量呢?
使用一般線性模型時,是需要滿足以下3點假設的:
正態性,因變量y符合正態分布
獨立性,不同類別y的觀察值之間相互獨立,相關係數為零
方差齊性,不同類別y的方差相等
以性別這個分類變量為例,如果不同性別對應的因變量值有明顯差異,也就說我們常說數據分層,那麽就不滿足上述條件了。此時如果堅持使用一般線性模型來擬合所有樣本,其參數估計值不在具有最小方差線性無偏性,回歸係數的標準誤差會被低估,利用回歸方程得到的估計值也會過高。
對於分層明顯的數據,一種解決方案就是將不同的層分開處理,比如性別分層,那麽就將不同性別的數據分開,每一類單獨處理,但是這要求每一類包含的樣本數據量要夠多,而且分層因素的類別也不能太多,太多了處理起來也很麻煩。另外一個解決方案就是更換模型,使用線性混合模型。
一般線性模型有3個前提條件,而線性混合模型隻保留了其中的第一點,即因變量要符合正態分布,對於獨立性和方差齊性不做要求,所以適用範圍更加廣泛。
在線性混合模型中,隨機效應變量Z的參數向量Γ服從均值為0,方差為G的正態分布,即Γ ~ N(0, G), 隨機誤差ε服從均值為,方差為R的正態分布,即ε ~ N(0, R), 同時假定G和R沒有相關性,即Cov(G, R) = 0, 此時因變量Y的方差可以表示為
Var(Y) = ZGZ + R
在GCTA軟件中,其核心就是線性混合模型,將所有SNP作為自變量,然後通過上述公式來估算SNP遺傳力。
對於一般線性模型,可以通過最小二乘法或者最大似然法來估算其參數,對於線性混合模型,常用的參數估方法為約束性最大似然法
restricted maximum likelihood 簡稱REML, 對於如下的混合模型:
其中y是已知的,表示因變量的觀測值,β是未知的,表示固定效應的參數向量,u是未知的,表示隨機效應的參數向量,對於該方程的參數估計,其實就是求解β和u的值,公式如下
對於固定效應β, 其估計值為最佳線性無偏估計 best linear unbiased estimates(BLUE)
對於隨機效應u, 其估計值為最佳線性無偏預測best linear unbiased predictors(BLUP)
線性混合模型隻要求因變量服從正態分布,適用範圍廣,在遺傳統計學中廣泛使用。
1.5.2 線性混合效應模型入門之一(linear mixed effects model)
縮寫LMM,在生物醫學或社會學研究中經常會用到。它主要適用於內部存在層次結構或聚集的數據,大體上有兩種情況:
(1)內部聚集數據:比如要研究A、B兩種教學方法對學生考試成績的影響,從4所學校選取1000名學生作為研究對象。由於學校之間的差異,來自其中某一所學校的學生成績可能整體都好於另一所學校,換句話說就是學生成績在學校這個維度上存在聚集現象。
(2)重複測量數據:比如要研究A、B兩種降壓藥物對高血壓患者血壓的影響,在每個患者服藥前、服藥後1個月、3個月、6個月分別測量血壓。由於同一個患者的每次血壓之間存在明顯的相關性,不能適用於傳統的方差分析方法。
隨機效應與固定效應
之所以稱為“線性混合效應模型”,就是因為這種模型結合了固定效應和隨機效應。
固定效應(fixed effect):所謂固定效應,指的是這個因素的每個水平(level)已經“窮舉”出來了,不能或者不需要再做“推廣”。比如上麵的降壓藥物研究,雖然降壓藥物有很多,但是研究者隻關心A、B兩種藥物的效果,所以可以視為固定效應。固定效應影響的是響應變量或因變量(如血壓)的均值。
隨機效應(random effect):指的是該因素是從一個更大的總體中抽取出來的樣本,我們的研究結果要推廣到整個總體。還是上麵的藥物研究,參與研究的患者隻是一個小樣本,所以患者作為隨機效應。隨機效應影響的是響應變量(血壓)的變異程度即方差。

https://www.nature.com/articles/nmeth.3137.epdf
圖a中演示是固定效應因子,每次重複實驗,因子都是A1、A2、A3三個水平,三個水平的效應均值是固定的。
圖b演示的是隨機效應因子,每次重複實驗,因子水平都不一樣,如第一次是B1、B2、B3,第二次是B4、B5、B6,以此類推。所以因子的每個水平對均值的影響都是隨機的,不固定的。
當然這兩種效應有時並不是絕對的,主要還是看研究的目的。
線性混合效應模型入門之二 - 實例操作及結果解讀(R、Python、SPSS實現
更新1:本文所用的數據下載地址: https://https://pan.baidu.com/s/1PYcMxDYgjjSvv9seUXmIDg?pwd=ngkw YgjjSvv9seUXmIDg?pwd=ngkw
更新2:評論區有人提出,R的結果和SPSS的結果不一致,這裏解釋一下,這是因為分類變量的類別參照不同,導致係數的符號相反,截距也不一樣。R用的是0值(gender=0,micro=0,macro=0)作為參照,而SPSS用的是第一個出現的類別(gender=1,micro=1,macro=1)作為參照。兩者結果數值不一樣,但是本質是一樣的。
--------------------------------------------------------------------------
下麵以一個具體的案例,說明線性混合效應模型的操作及結果解讀,本文以三種方式進行實現:分別是Python、R、SPSS。
案例數據介紹
本案例數據來源於一個腎髒病的研究。研究對200個腎病患者進行隨訪,每年化驗一次腎小球濾過率(GFR,評價腎髒功能的指標,會逐年下降)。主要分析目的是探索基線的尿蛋白定量對GFR年下降率(斜率)的影響(尿蛋白量越大,對腎功能危害越大),混雜因素包括基線年齡和性別。
數據展示如下:
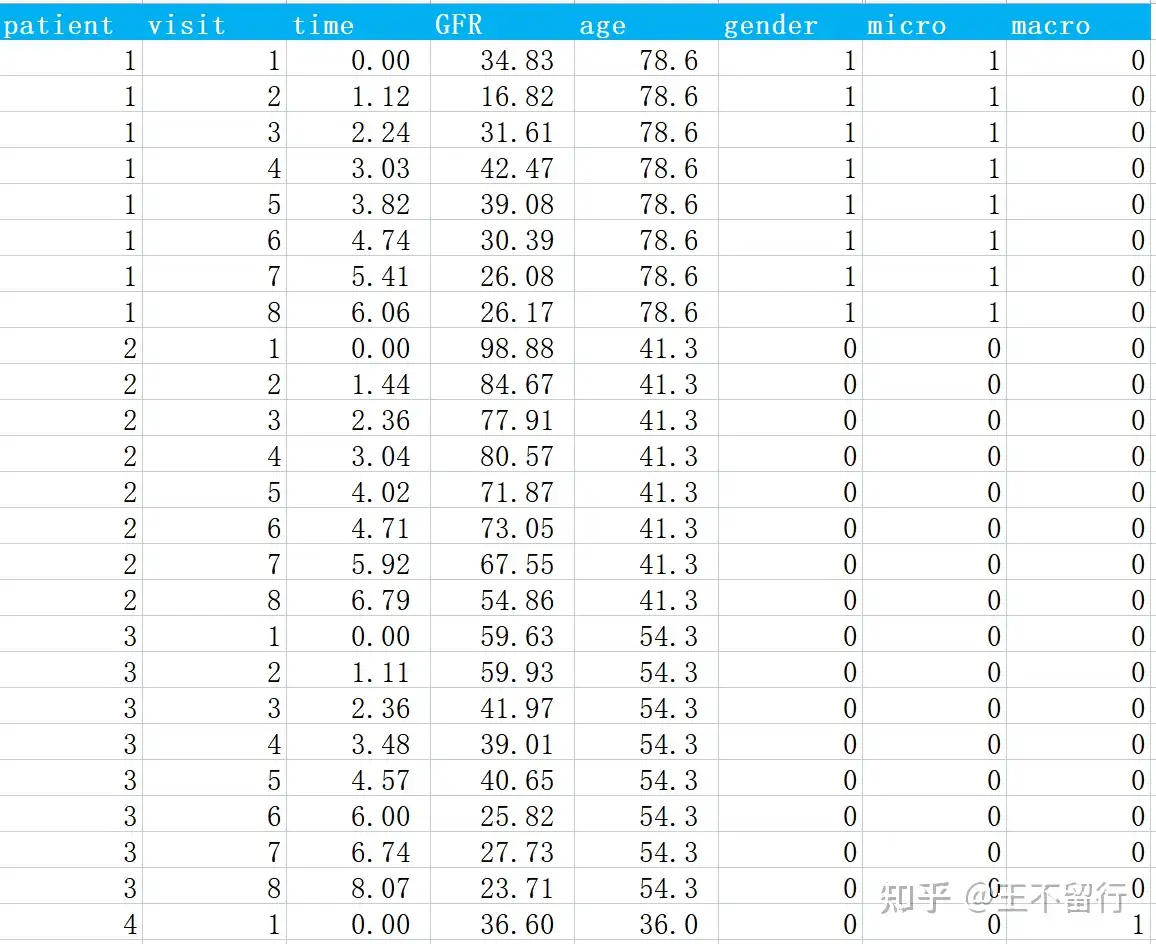
字段說明:
(1)patient: 患者ID編號;
(2)visit:化驗次序編號;
(3)time:化驗時間(單位年),第一次化驗定為0,後麵依次推延;
(4)GFR:腎小球濾過率,單位是ml/min/1.73^2,作為響應變量;
(5)age:基線年齡,單位歲;
(6)gender:性別,0=男,1=女;
(7)micro:基線是否有微量蛋白尿,0=無,1=有;
(8)macro:基線是否有大量蛋白尿,0=無,1=有;
補充說明:
(1)蛋白尿這裏用了啞變量編碼,macro=0且micro=0表示沒有蛋白尿;
(2)數據中GFR化驗數據有缺失,線性混合效應模型對缺失數據有良好的處理能力。
統計模型構建
由於一個患者進行了多次的GFR化驗檢查,屬於重複測量數據,GFR不符合隨機獨立的要求。所以采用線性混合模型來建模。這裏的響應變量是GFR,預測變量是基線年齡、性別、尿蛋白情況、化驗時間、尿蛋白情況,其中尿蛋白是主要關注的因子。根據專業醫學知識,我們假設尿蛋白不僅影響GFR的下降率,也影響基線GFR。所以預測變量還需要加上一個時間x尿蛋白的交互項(這裏交互項的意思是說,不同的尿蛋白等級會有不同的GFR下降斜率和下降曲線)。另外假設性別、年齡僅影響基線的GFR,不影響GFR下降率(這個假設不一定合理,這裏是為了模型簡化,如果假設不成立,需要和尿蛋白一樣乘以時間交互項)。用公式表示如下:
β1 等於基線時刻(time=0),在age和gender相同的情況下,微量蛋白組(micro)和正常蛋白組(micro=0且macro=0)人群基線GFR的均值的差值。
β2 等於基線時刻(time=0),在age和gender相同的情況下,大量蛋白組(macro)和正常蛋白組(micro=0且macro=0)人群基線GFR的均值的差值。
β3 等於正常尿蛋白組(micro=0且macro=0)人群GFR斜率的均值。
β4 等於微量蛋白組(micro)和正常蛋白組(micro=0且macro=0)GFR斜率均值的差值。
β5 等於大量蛋白組(macro)和正常蛋白組(micro=0且macro=0)GFR斜率均值的差值。
β3+β4 等於微量蛋白組(micro)人群GFR斜率的均值。
β3+β5 等於大量蛋白組(macro)人群GFR斜率的均值。
u0i 等於第i個患者和相同基線特征人群的基線GFR均值的隨機偏差,服從均值為零和同方差的獨立正態分布。
u1i 等於第i個患者和人群GFR斜率均值(如β3,β3+β4,β3+β5)的隨機偏差,服從均值為零和同方差的獨立正態分布。
eij 等於TIMEij時間的隨機誤差,服從均值為零和同方差的獨立正態分布。
對照上文,我們就可以從統計軟件得出的結果中找出對應的感興趣的數據,進行相應的解釋。
對照上文,我們就可以從統計軟件得出的結果中找出對應的感興趣的數據,進行相應的解釋。
統計實現
1、R語言實現
核心代碼:
library(nlme) # Fit Gaussian linear and nonlinear mixed-effects models
library(lme4) # Fit linear and generalized linear mixed-effects models
library(epiR) # Analysis of epidemiological data
library(epicalc) # Functions for epidemiological calculations
library(lattice) # Data visualization system
library(epiDisplay)
## IMPORT DATA
#-------------------------------
dropout.dat <- read.table("./data_dropout.csv", sep = ",", na.string = " ", header = TRUE, dec = ".")
## Linear mixed model
#-------------------------------
cat("LINEAR MIXED MODEL WITH SYMMETRIC COVARIANCE MATRIXn")
fit.m3 <- lme(GFR ~ time + age + gender + micro + macro + micro:time + macro:time,
random = list(patient = pdSymm( ~ 1 + time)), method = "ML",
data = dropout.dat, control = lmeControl(opt = "optim"))
# random argument: is identical to random = ~ 1 + time|patient
summary(fit.m3)
cat("95% CIn")
coef.fit.m3 <- summary(fit.m3)$tTable
ci(coef.fit.m3[,1], coef.fit.m3[,2], coef.fit.m3[,4])
cat("VARIANCE-COVARIANCE MATRIXn")
VarCorr(fit.m3)
輸出結果:
> summary(fit.m3)
Linear mixed-effects model fit by maximum likelihood
Data: dropout.dat
AIC BIC logLik
9540.1 9602.84 -4758.05
Random effects:
Formula: ~1 + time | patient
Structure: General positive-definite
StdDev Corr
(Intercept) 10.426283 (Intr)
time 3.591427 -0.053
Residual 4.903330
Fixed effects: GFR ~ time + age + gender + micro + macro + micro:time + macro:time
Value Std.Error DF t-value p-value
(Intercept) 83.39609 3.242467 1175 25.719948 0.0000
time -1.74102 0.414817 1175 -4.197072 0.0000
age -0.28739 0.051564 195 -5.573483 0.0000
gender -2.61848 1.682065 195 -1.556703 0.1212
micro -18.39949 1.876851 195 -9.803381 0.0000
macro -26.56472 1.898283 195 -13.994078 0.0000
time:micro -2.92088 0.638801 1175 -4.572445 0.0000
time:macro -3.09101 0.670447 1175 -4.610374 0.0000
Correlation:
(Intr) time age gender micro macro time:micr
time -0.039
age -0.914 0.001
gender -0.018 0.000 -0.136
micro -0.169 0.067 -0.085 -0.002
macro -0.244 0.066 0.014 -0.108 0.425
time:micro 0.026 -0.649 -0.001 -0.002 -0.113 -0.043
time:macro 0.023 -0.619 0.001 -0.005 -0.041 -0.123 0.402
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-3.16590972 -0.60414300 -0.02061946 0.58097222 3.14244493
Number of Observations: 1378
Number of Groups: 200
> coef.fit.m3 <- summary(fit.m3)$tTable
> ci(coef.fit.m3[,1], coef.fit.m3[,2], coef.fit.m3[,4])
beta ci.low ci.up pvalue
(Intercept) 83.40 77.04 89.75 0.0000
time -1.74 -2.55 -0.93 0.0000
age -0.29 -0.39 -0.19 0.0000
gender -2.62 -5.92 0.68 0.1195
micro -18.40 -22.08 -14.72 0.0000
macro -26.56 -30.29 -22.84 0.0000
time:micro -2.92 -4.17 -1.67 0.0000
time:macro -3.09 -4.41 -1.78 0.0000
>
> cat("VARIANCE-COVARIANCE MATRIXn")
VARIANCE-COVARIANCE MATRIX
> VarCorr(fit.m3)
patient = pdSymm(1 + time)
Variance StdDev Corr
(Intercept) 108.70738 10.426283 (Intr)
time 12.89835 3.591427 -0.053
Residual 24.04265 4.903330
根據輸出結果,我們整理成下表:
| 指標 | 估計值(95% CI) |
|---|---|
| 1、基線GFR差值(校正性別、年齡) | |
| (1)微量蛋白組(micro)-正常蛋白組差值 | -18.4(-22.1, -14.7) |
| (2)大量蛋白組(macro)-正常蛋白組差值 | -26.6(-30.3, -22.9) |
| 2、平均GFR年下降率(斜率) | |
| (1)正常蛋白組 | -1.74(-2.55, -0.93) |
| (2)微量蛋白組(micro)-正常蛋白組差值 | -2.92(-4.17, -1.67) |
| (3)大量蛋白組(macro)-正常蛋白組差值 | -3.09(-4.41, -1.78) |
2、SPSS 實現

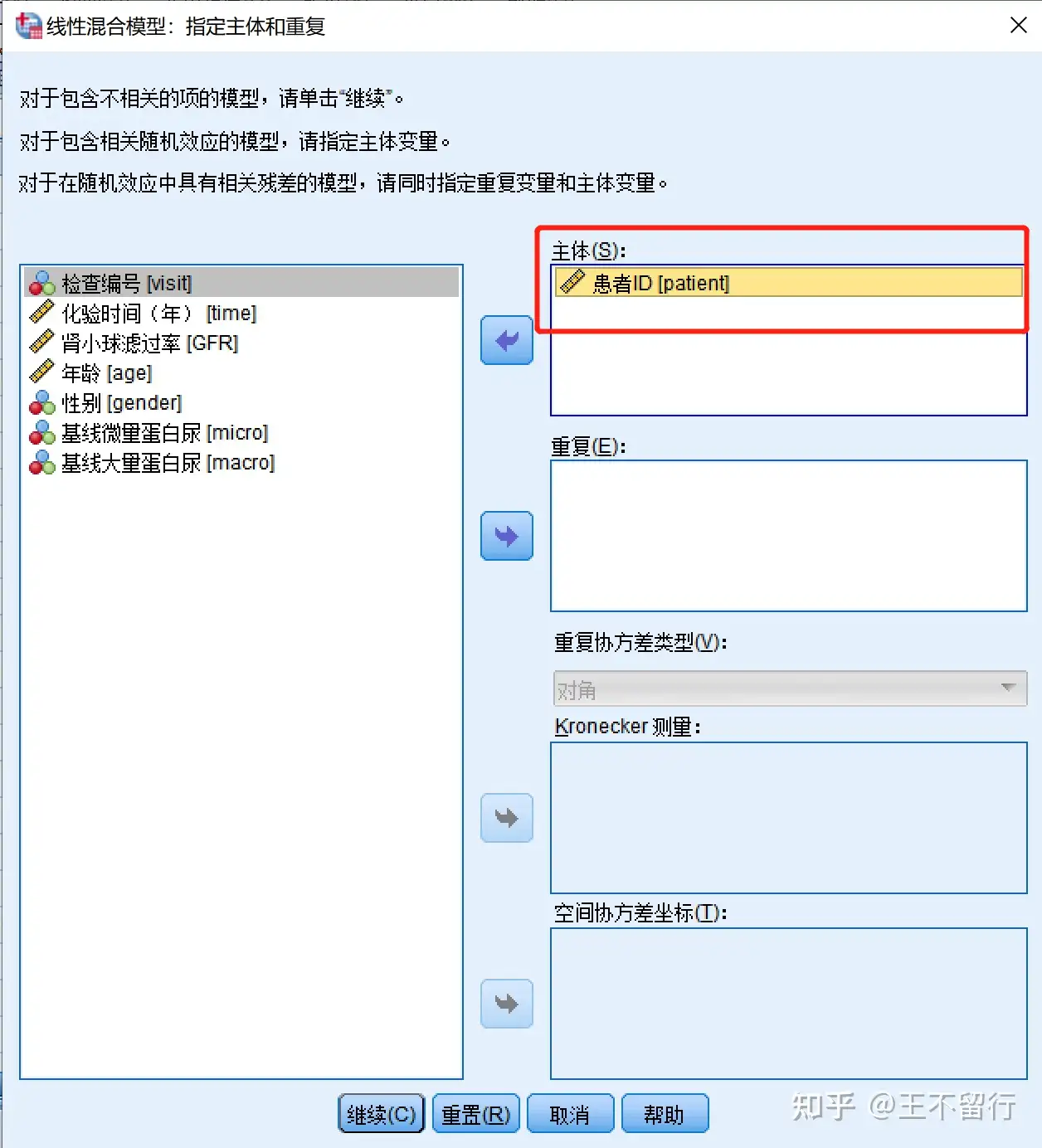
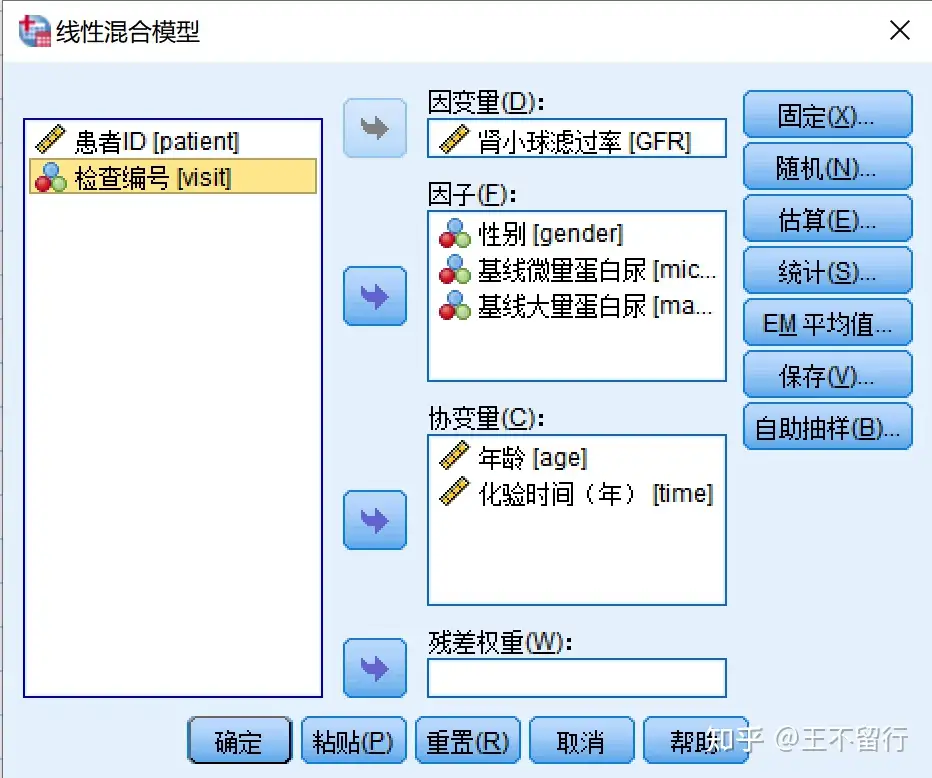
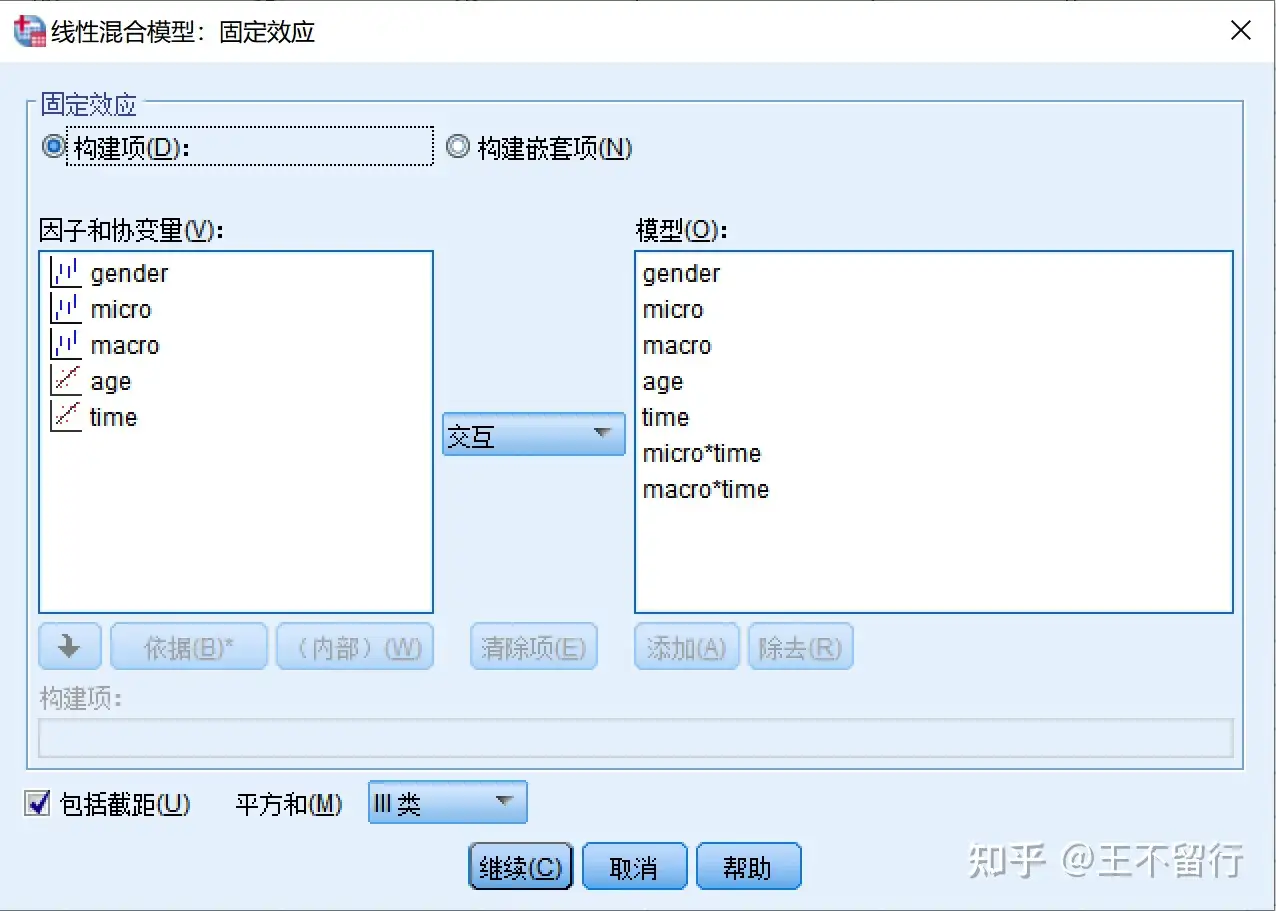
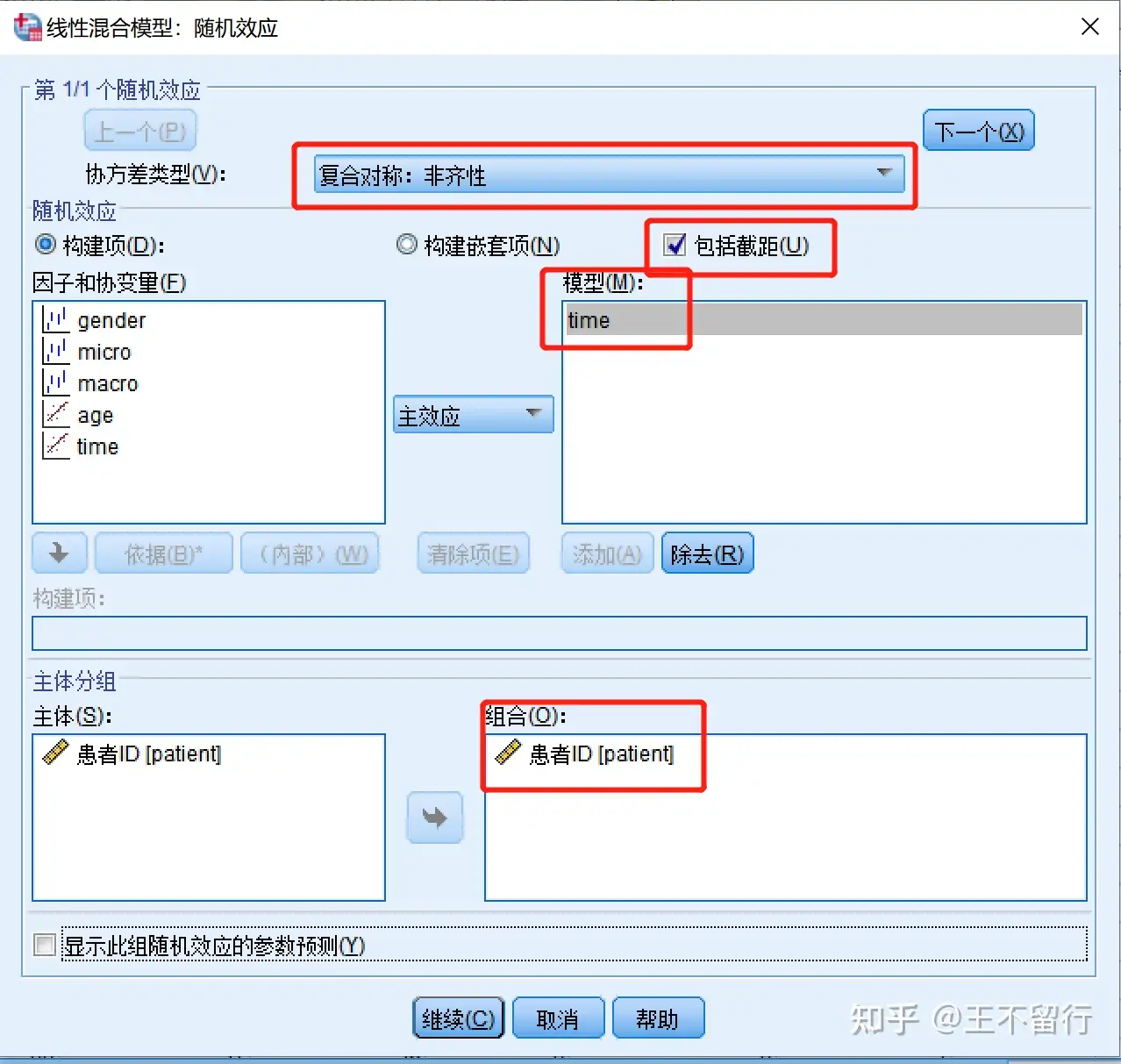

mango:此論文丟了一步即勾選統計量,點擊Ok即可在左欄觀察結果。SPSS9.2下得出的結果類似下述,估計值類似但標準差小些。
輸出結果:
結果與R語言的一致。但是由於SPSS默認選擇的分類變量參照不同,導致係數的符號相反,截距數值也不一樣,本質是一樣的。
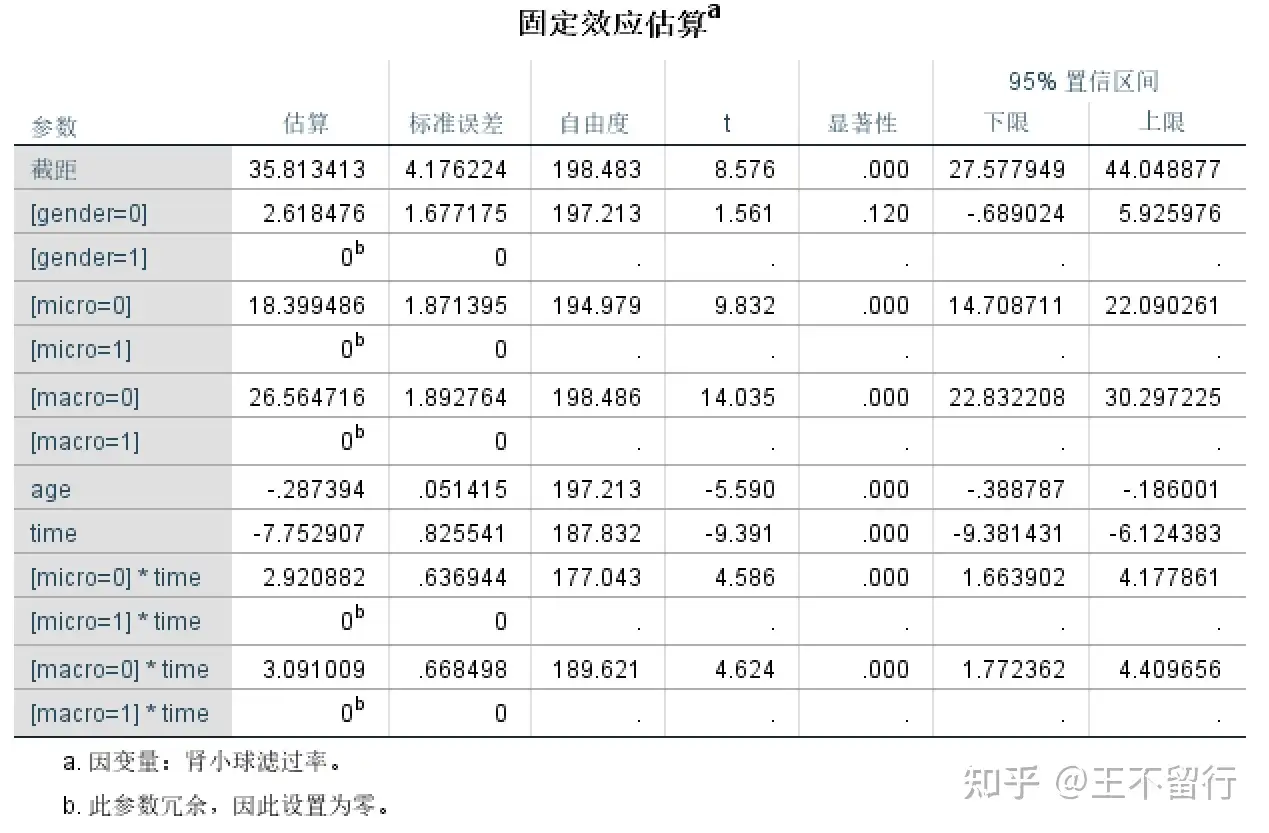
3、Python實現
Python的statsmodels包可以計算混合模型,但是計算開銷比較大。可以在本地安裝R環境,同時安裝rpy2和pymer4,就可以用python調用R,非常方便。核心代碼如下:
import pandas as pd
from pymer4.models import Lmer
data = pd.read_csv("./data_dropout.csv")
model = Lmer(formula="GFR ~ time + age + gender + micro + macro + micro:time + macro:time + (1 + time | patient)",
data=data)
model.fit()
print(model.summary())
輸出結果:
Formula: GFR~time+age+gender+micro+macro+micro:time+macro:time+(1+time|patient)
Family: gaussian Inference: parametric
Number of observations: 1378 Groups: {'patient': 200.0}
Log-likelihood: -4754.489 AIC: 9508.979
Random effects:
Name Var Std
patient (Intercept) 111.803 10.574
patient time 13.130 3.624
Residual 24.038 4.903
IV1 IV2 Corr
patient (Intercept) time -0.054
Fixed effects:
Formula: GFR~time+age+gender+micro+macro+micro:time+macro:time+(1+time|patient)
Family: gaussian Inference: parametric
Number of observations: 1378 Groups: {'patient': 200.0}
Log-likelihood: -4754.489 AIC: 9508.979
Random effects:
Name Var Std
patient (Intercept) 111.803 10.574
patient time 13.130 3.624
Residual 24.038 4.903
IV1 IV2 Corr
patient (Intercept) time -0.054
Fixed effects:
Estimate 2.5_ci 97.5_ci SE DF T-stat P-val Sig
(Intercept) 83.402 76.984 89.820 3.275 192.390 25.469 0.000 ***
time -1.741 -2.559 -0.923 0.417 165.191 -4.174 0.000 ***
age -0.288 -0.390 -0.185 0.052 192.333 -5.521 0.000 ***
gender -2.616 -5.945 0.714 1.699 192.332 -1.540 0.125
micro -18.396 -22.111 -14.681 1.895 190.263 -9.705 0.000 ***
macro -26.565 -30.322 -22.808 1.917 193.610 -13.858 0.000 ***
time:micro -2.924 -4.182 -1.665 0.642 174.480 -4.552 0.000 ***
time:macro -3.096 -4.417 -1.775 0.674 186.786 -4.594 0.000 ***
2. SAS studio
由此:https://www.sas.com/profile/ui/#/sign-in 注冊SAS賬號,收到SAS賬戶激活郵件,激活賬號並設置密碼後,來到SAS Studio官網激活SAS OnDemand for Academics賬戶的用戶ID,該用戶ID會在後麵用來登陸SAS Studio。登錄:https://welcome.oda.sas.com u63668635
我們在程序標簽頁下運行「Proc setinint; run;」看看許可了哪些模塊,結果發現除了常規的Base SAS、SAS/STAT、SAS/GRAPH、SAS EnterpriseGuid外,SAS/IML、SAS/ETS、 SAS/OR、 SAS/QC、SAS/CONNECT等模塊也都赫然在列, 甚至連數據挖掘和文本挖掘的產品SAS Enterprise Miner 、SAS TextMiner以及可視化分析產品SAS Visual AnalyticsHub、Visual Analytics Explorer 、SAS Visual Analytics Services 都囊括在其中,不得說,SAS公司誠意滿滿啊。




